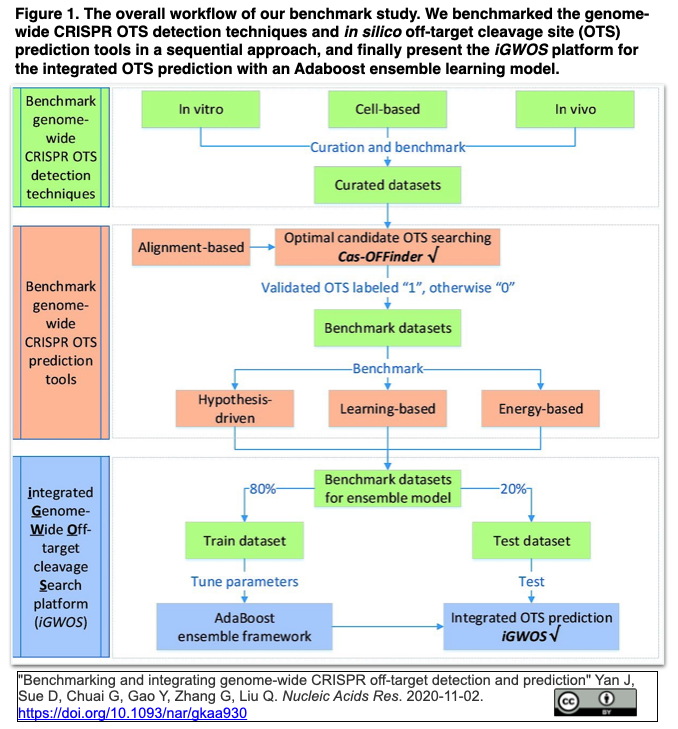
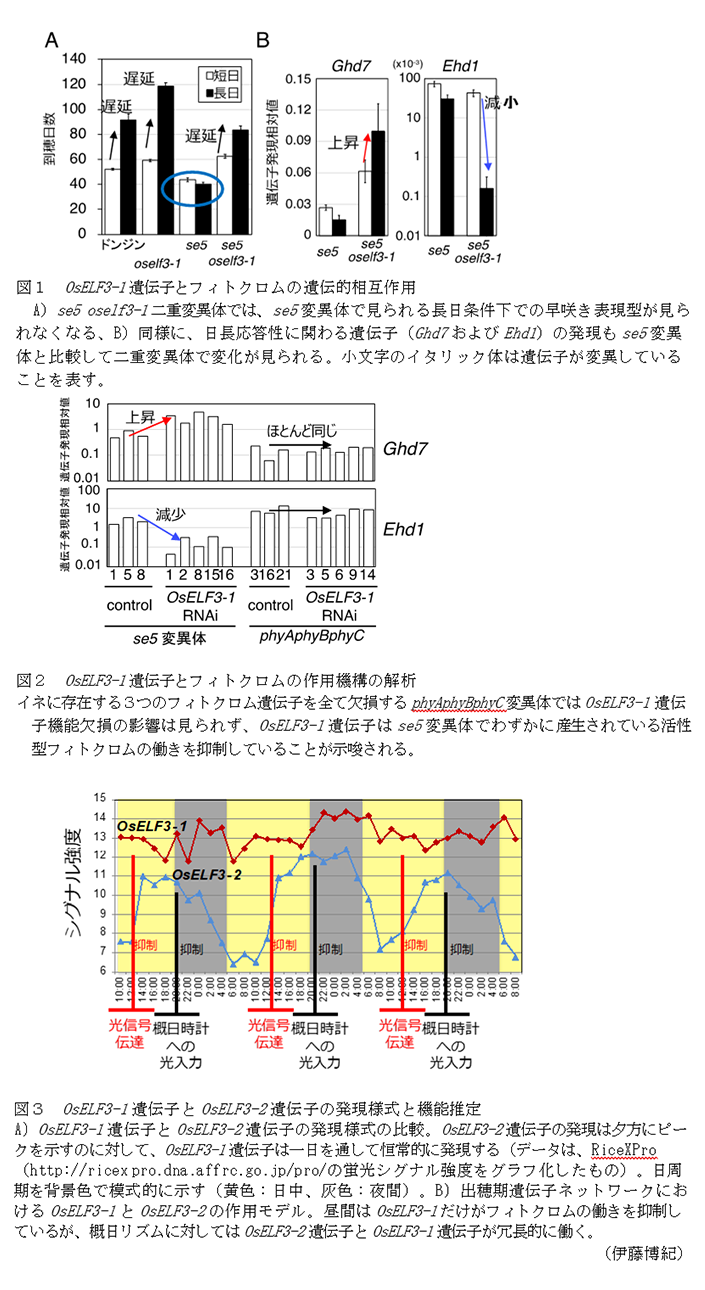
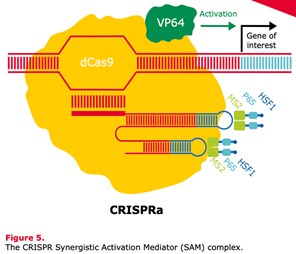
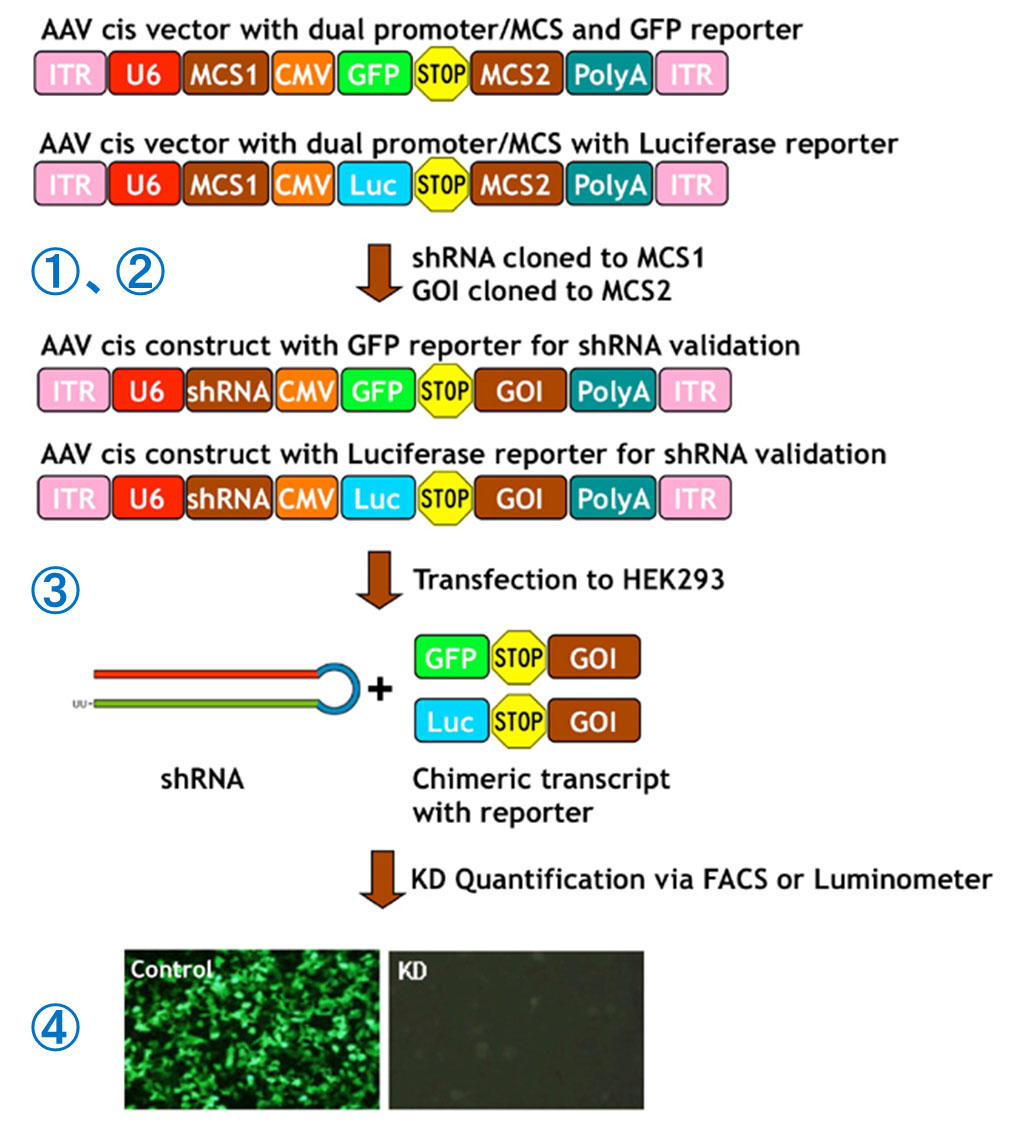
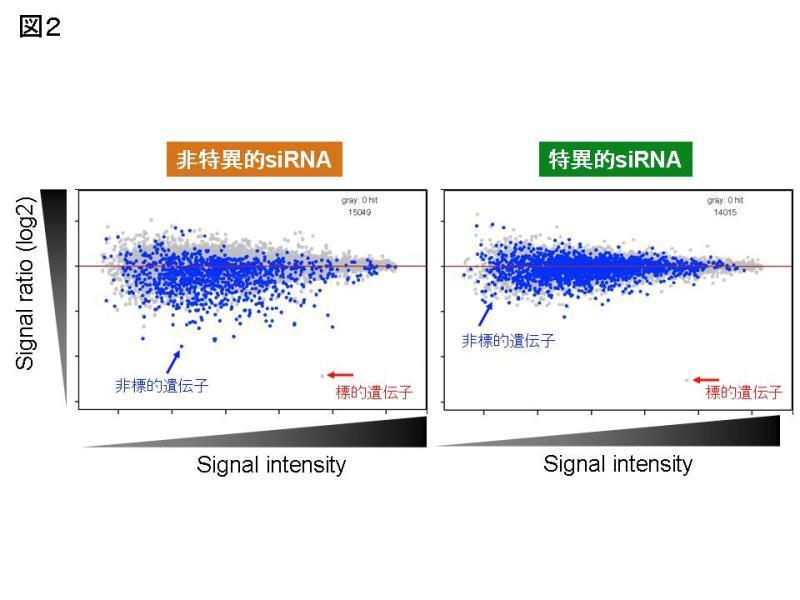
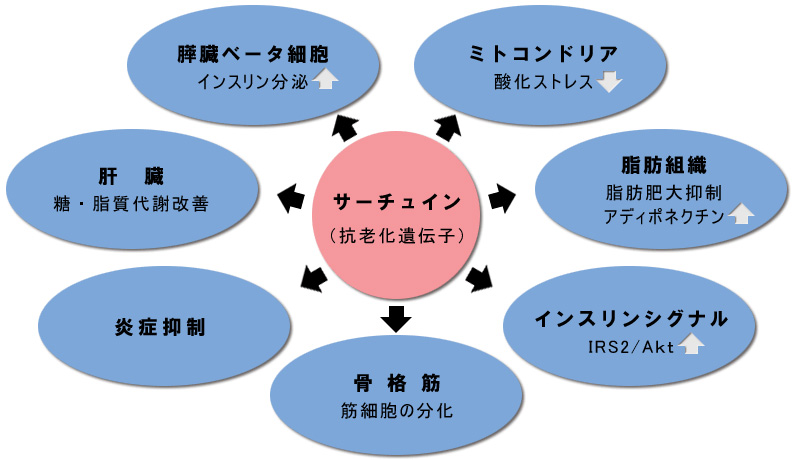
Images of 遺伝子サイレンシング

松永政司公式ブログ ~生活習慣の改善で、健康・長寿社会の実現を!~2019年06月核酸サプリメント愛用者の皆様へ(その17)核酸サプリメント愛用者の皆様へ(その16)核酸サプリメント愛用者の皆様へ(その15)
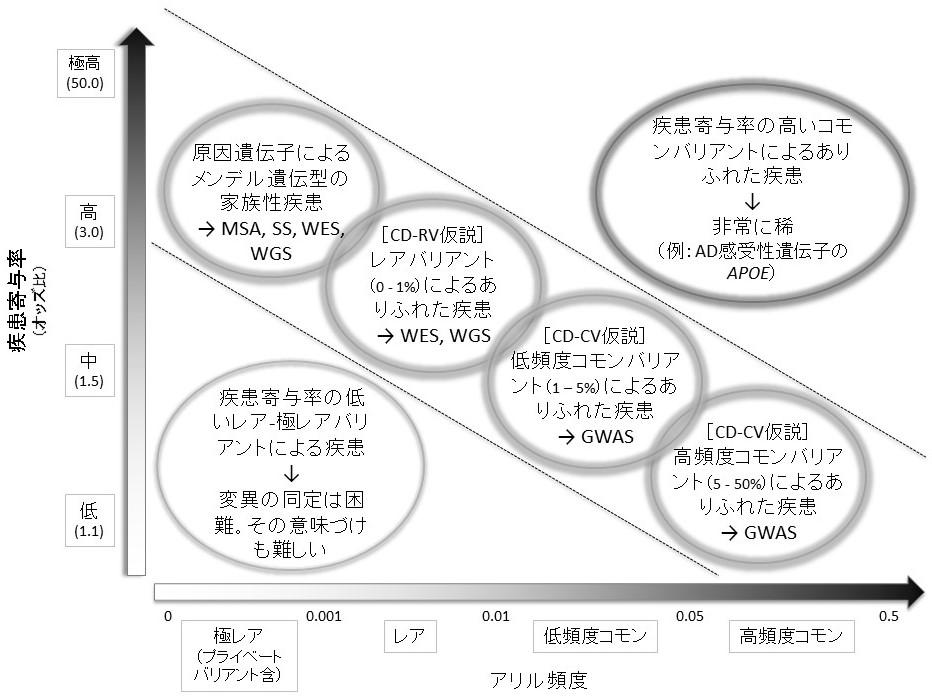
アルツハイマー病の原因遺伝子、感受性遺伝子(2018年9月3日公開)図1 | 疾患の原因・感受性遺伝子探索におけるアリル頻度と疾患寄与率の関係文献17のボックス7と文献18の図1をもとに作成した。メンデル遺伝型の家族性疾患の原因遺伝子は、マイクロサテライトマーカーによる連鎖解析とポジショナルクローニングによって、主に1980-90年代に同定された。また、近年ではNGSを駆使した全ゲノム・エクソンシーケンシングにより、原因遺伝子が同定されている。アリルの頻度は非常に稀であるが、その疾患寄与率は非常に高い(ほぼ100%)。2000年代に入りコモンバリアントであるSNPベースのGWASが行われ、がん、糖尿病、高血圧、ADなどのありふれた疾患の感受性遺伝子が見出された。疾患に寄与するアリルの頻度は高いものの(1%以上)、その大きさは低い(オッズ比はおよそ1.5未満)。2010年代に入り、NGSによるありふれた疾患のレアバリアント探索が活発化した。民族固有の、あるいは家系固有の感受性遺伝子が相次いで報告されている。アリルの頻度は低い(1%未満)ながらもその疾患寄与率は高い(オッズ比2以上)。NGSをもってしても孤発性のありふれた疾患に関与する極レアバリアントやプライベートバリアントの同定は非常に難しく、その意味づけも困難である。一方、高頻度のコモンバリアント(5%以上)が高い疾患寄与率(オッズ比3以上)を呈してありふれた疾患に関連する例は非常に稀だが、ADの感受性遺伝子であるAPOEはそれに相当する。図2 | 欧米人による大規模メタGWASによって認められたAD感受性遺伝子文献9の図1を改変した。欧米人のAD17,008例と対照37,154例を用いたステージ1の解析のマンハッタンプロット。既報のGWASで同定されていた遺伝子は黒字(CR1、BIN1、CD2AP、EPHA1、CLU、MS4A6A、PICALM、ABCA7、APOE、CD33)、新たに見出された遺伝子を青字(ステージ1の新規遺伝子:HLA-DRB5-DRB1、PTK2B、SORL1、SLC24A4-RIN3、DSG2)と赤字(ステージ1+2の新規遺伝子:INPP5D、MEF2C、NME8、ZCWPW1、CELF1、FERMT2、CASS4)でそれぞれ示す。最強のAD感受性遺伝子APOEは緑色の丸で囲んだ。APOE周辺領域では複数のSNPが強力に相関することが分かる。SORL1は我々のGWAS(文献10)でも同定されたので紫色の四角で囲み強調した。図3 | ADの原因・感受性遺伝子文献19の図1を改変した。欧米人による遺伝学的解析で見出されたADの原因・感受性遺伝子の集団内頻度とリスクレベルとの関係を示す。各遺伝子は機能に応じて色分けされている。†, TREM2の解析(文献14)に使用した検体(臨床+病理検体:疾患群2,190例、対照群2,498例)で算出した。APOE-ε3*3に対するAPOE-ε4*4(28.28)とAPOE-ε3*4(4.87)のオッズ比を示す。*, TREM2は日本人を含む東アジア人のADとは相関しない。$, 我々の報告したSORL1のSNP rs3781834のオッズ比(0.74)を示す(文献10)。(略語)AD, Alzheimer's diseaseAβ, amyloid betaCD, common diseaseCV, common variantGWAS, genome-wide association studyMSA, microsatellite maker analysisNGS, next generation sequencer/sequencingRV, rare variantSNP, single nucleotide polymorphismSS, Sanger sequencingWES, whole-exome sequencingWGS, whole-genome sequencing(文献)1. Alzheimer's Disease International: The World Alzheimer Report 2015, The Global Impact of Dementia: An analysis of prevalence, incidence, cost and trends (https://www.alz.co.uk/research/WorldAlzheimerReport2015.pdf)2. Selkoe DJ, Hardy J: The amyloid hypothesis of Alzheimer's disease at 25 years. EMBO Mol Med, 8:595-608 (2016).3. Kasuga K, Kikuchi M, Tokutake T, Nakaya A, et al.: Systematic review and meta-analysis of Japanese familial Alzheimer's disease and FTDP-17. J Hum Genet, 60:281-283 (2015).4. Corbo RM1, Scacchi R: Apolipoprotein E (APOE) allele distribution in the world. Is APOE*4 a 'thrifty' allele? Ann Hum Genet, 63:301-310 (1999).5. Seripa D, Matera MG, Daniele A, Bizzarro A, et al.: The missing ApoE allele. Ann Hum Genet, 71:496-500 (2007).6. Namba Y, Tomonaga M, Kawasaki H, Otomo E, et al: Apolipoprotein E immunoreactivity in cerebral amyloid deposits and neurofibrillary tangles in Alzheimer's disease and kuru plaque amyloid in Creutzfeldt-Jakob disease. Brain Res, 541:163-166 (1991).7. 徳武孝允, 春日健作, 原範和, 池内健:アポリポ蛋白E遺伝子(APOE). BRAIN and NERVE, 68(7): 703 - 712 (2016).8. Coon KD, Myers AJ, Craig DW, Webster JA, et al.: A high-density whole-genome association study reveals that APOE is the major susceptibility gene for sporadic late-onset Alzheimer's disease. J Clin Psychiatry, 68:613-618 (2007).9. Lambert JC, Ibrahim-Verbaas CA, Harold D, Naj AC, et al.: Meta-analysis of 74,046 individuals identifies 11 new susceptibility loci for Alzheimer's disease. Nat Genet, 45:1452-1458 (2013).10. Miyashita A, Koike A, Jun G, Wang LS, et al.: SORL1 is genetically associated with late-onset Alzheimer's disease in Japanese, Koreans and Caucasians. PLoS One, 8:e58618 (2013).11. Hirano A, Ohara T, Takahashi A, Aoki M, et al.: A genome-wide association study of late-onset Alzheimer's disease in a Japanese population. Psychiatr Genet, 25:139-146 (2015).12. Holstege H, van der Lee SJ, Hulsman M, Wong TH, et al.: Characterization of pathogenic SORL1 genetic variants for association with Alzheimer's disease: a clinical interpretation strategy. Eur J Hum Genet, 25:973-981 (2017).13. Sims R, van der Lee SJ, Naj AC, Bellenguez C, et al.: Rare coding variants in PLCG2, ABI3, and TREM2 implicate microglial-mediated innate immunity in Alzheimer's disease. Nat Genet, 49:1373-1384 (2017).14. Miyashita A, Wen Y, Kitamura N, Matsubara E, et al.: Lack of genetic association between TREM2 and late-onset Alzheimer's disease in a Japanese population. J Alzheimers Dis, 41:1031-1038 (2014).15. Escott-Price V, Sims R, Bannister C, Harold D, et al.: Common polygenic variation enhances risk prediction for Alzheimer's disease. Brain, 138:3673-3684 (2015).16. Desikan RS, Fan CC, Wang Y, Schork AJ, et al.: Genetic assessment of age-associated Alzheimer disease risk: Development and validation of a polygenic hazard score. PLoS Med, 14:e1002258 (2017).17. McCarthy MI, Abecasis GR, Cardon LR, Goldstein DB, et al.: Genome-wide association studies for complex traits: consensus, uncertainty and challenges. Nat Rev Genet, 9:356-369 (2008). 18. Manolio TA, Collins FS, Cox NJ, Goldstein DB, et al.: Finding the missing heritability of complex diseases. Nature, 461:747-753 (2009).19. Karch CM1, Goate AM: Alzheimer's disease risk genes and mechanisms of disease pathogenesis. Biol Psychiatry, 77:43-51 (2015).20. 宮下哲典, 原範和, 春日健作, 菊地正隆, 他:アルツハイマー病の遺伝学的リスク. 老年精神医学雑誌, 28(7): 754-765 (2017).





















![[P20倍/28日まで] [レビュー11000件超え/高評価4.59点] 毛布 シングル 140×200cm ブランケット 冬 ひざ掛け 膝掛け 掛け毛布 160×200cm 180×200cm レギュラー ボリュームタイプ 4層プレミアム マイクロファイバー フランネル セミダブル ダブル 暖かい](https://thumbnail.image.rakuten.co.jp/@0_mall/dondon/cabinet/zmf01_3.jpg?_ex=300x300)



![2大特典[ 分別ダストワゴン タワー / 蓋付き / 目隠し / 2分別 / 3分別 / 45L ]山崎実業 tower キッチン ゴミ箱 ごみ箱 ダストボックス ゴミ袋ハンガー フタ付き ふた 分別 ゴミ袋ホルダー キャスター付き レジ袋ホルダー 大容量 黒 白 おしゃれ カウンター下 北欧 公式](https://thumbnail.image.rakuten.co.jp/@0_mall/roomy/cabinet/500cart_all/500cart_11g/p5n-7/ymz1002-st019-0_gt01.jpg?_ex=300x300)